Análisis de imágenes (bio) con Python Leer y cargar imágenes microscópicas utilizando Matplotlib
Análisis de imágenes (bio) con Python utilizando Matplotlib
En las últimas dos décadas, el campo de la microscopía de luz ha presenciado avances notables gracias a la introducción de técnicas revolucionarias como la microscopía de barrido láser confocal (CLSM), la microscopía de barrido láser de fotones múltiples (MPLSM), la microscopía de fluorescencia de hoja de luz (LSFM) y la microscopía de superresolución [1]. Estas técnicas han mejorado significativamente la capacidad de capturar imágenes con una mayor penetración en la profundidad, viabilidad y resolución. Sin embargo, junto con estos avances notables, la aparición de estas técnicas sofisticadas ha presentado desafíos considerables en el manejo de las grandes cantidades de datos generados. La visualización, análisis y difusión efectivos de estos conjuntos de datos complejos se han convertido en un aspecto crucial para aprovechar todo el potencial de estas técnicas de microscopía de vanguardia. En este tutorial, exploraremos cómo leer y visualizar imágenes de microscopía de luz 2D, 3D, 4D y 5D complejas utilizando la potente biblioteca de Python Matplotlib.
Tabla de contenidos
- 1.1 Imágenes y píxeles
- 1.2 Canales
- 1.3 Imágenes 4D (x, y, canales y z-slices)
- 1.4 Proyecciones
- 1.5 Imágenes 5D (x, y, canales, z-slices y tiempo)
- 1.6 Conclusiones
- 1.7 Referencias
# Importar bibliotecasimport numpy as npimport matplotlib.pyplot as pltimport skimage as skfrom matplotlib_scalebar.scalebar import ScaleBarImágenes y píxeles
Las imágenes microscópicas se registran digitalmente y, para lograr esto, el flujo de fotones que forma la imagen final debe dividirse en pequeñas subunidades geométricas, los elementos de imagen (píxeles) [2]. En lo que respecta a la computadora, cada píxel es solo un número (Fig. 1), y cuando se muestra los datos de la imagen, los valores de los píxeles generalmente se convierten en cuadrados; los cuadrados no son más que una visualización útil que nos permite obtener una impresión rápida del contenido de la imagen [3].
![Figura 1. Imagen microscópica de la localización subcelular de la proteína eisosomal PilA-GFP [5], [6] en el conidiosporo del hongo *Aspergillus nidulans* con un área ampliada (inserción roja) que muestra los valores de los píxeles. La imagen fue capturada utilizando un microscopio de multiphoton Leica TCS SP8 MP (figura proporcionada por el autor).](https://miro.medium.com/v2/format:webp/1*L4Luyzm04ji5moFMT36iEw.png)
En microscopía y específicamente en fluorescencia, las imágenes microscópicas generalmente se adquieren sin información de color (usando cámaras monocromáticas o fotomultiplicadores) y se visualizan utilizando una LUT de falso color monocromático. Las tablas de búsqueda (LUTs; a veces también llamadas mapas de color) determinan cómo se traducen los valores de gris en un valor de color. Para visualizar imágenes microscópicas, a menudo usamos tonos de gris (LUT gris), o en caso de que queramos aumentar la sensibilidad visual del lector a características tenues, invertimos la imagen, mapeando valores oscuros a claros y viceversa [4].
Todas las imágenes microscópicas publicadas deben contener una barra de escala como referencia visual para el tamaño de las estructuras o características observadas en la imagen.
- Cómo ajustar finamente un LLM para una tarea de preguntas y respuestas (QA) localmente
- GPT4Readability Nunca vuelvas a escribir un README de nuevo
- Liberando la creatividad Explorando las aplicaciones artísticas de la IA generativa
# Leer la imagen usando scikit-imageimg = sk.io.imread("Single_channel_eisosome.tif")# Imprimir las dimensiones de la imagenprint(f"Las dimensiones de las imágenes son: {img.shape[0]} x {img.shape[1]} píxeles")# Crear una figura con dos subtramasfig, axs = plt.subplots(1, 2, figsize=(6, 4))# Graficar la imagen en escala de grises en la primera subtramaaxs[0].imshow(img, cmap="gray")axs[0].axis('off')# Agregar una barra de escala a la subtramascalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')axs[0].add_artist(scalebar)axs[0].set_title("Imagen en escala de grises")# Graficar la imagen en escala de grises invertida en la segunda subtramaaxs[1].imshow(img, cmap="gray_r")axs[1].axis('off')# Agregar una barra de escala a la subtramascalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')axs[1].add_artist(scalebar)axs[1].set_title("Imagen invertida")# Ajustar el espaciado entre las subtramasplt.tight_layout()Las dimensiones de las imágenes son: 589 x 589 píxeles
Canales
En microscopía, diferentes canales representan distintos rangos espectrales utilizados para capturar y visualizar componentes o estructuras específicas en la muestra. Cada canal corresponde a un rango de longitud de onda específico o a un espectro de emisión, lo que permite a los investigadores visualizar simultáneamente diferentes moléculas, estructuras o eventos dentro de la muestra. Para optimizar el reconocimiento visual, se recomienda el uso de canales separados en blanco y negro para la mayoría de las aplicaciones de imagen fluorescente. Se puede crear una imagen final fusionada en color para mostrar la superposición, asegurando así la accesibilidad para lectores con formas comunes de daltonismo. En el código a continuación, visualizamos la localización subcelular de PilA-GFP (usando una tabla de búsqueda en escala de grises) y Histone H1-mRFP (usando una tabla de búsqueda en escala de grises), así como su superposición (PilA en magenta e Histone H1 en verde) [7] en el conidiósporo de A. nidulans.
# Leer la imagen usando scikit-imageimg = sk.io.imread("3_channel_eisosome.tif")# Imprimir las dimensiones de la imagenprint(f"Las dimensiones de las imágenes son {img.shape[0]} x {img.shape[1]} píxeles con {img.shape[2]} canales ")# Separar los canalesh1_histone = img[:,:,0]pila = img[:,:,1]brightfield = img[:,:,2]# Crear una figura con cuatro subplotsfig, axs = plt.subplots(1, 4, figsize=(12, 4))# Graficar H1 histone en el primer subplotaxs[0].imshow(h1_histone,cmap="gray")axs[0].axis('off')axs[0].set_title("H1 histone (Imagen en escala de grises)")# Graficar PilA en el segundo subplotaxs[1].imshow(pila,cmap="gray")axs[1].axis('off')axs[1].set_title("Proteína PilA (Imagen en escala de grises)")# Graficar la imagen brightfield en el tercer subplot con la escala axs[2].imshow(brightfield,cmap="gray")axs[2].axis('off')scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')axs[2].add_artist(scalebar)axs[2].set_title("Brightfield")# Convertir las rebanadas 2D en arreglos 3Dh1_histone_3d = np.atleast_3d(img[:,:,0]) #convierte la rebanada 2D en un arreglo 3Dpila_3d = np.atleast_3d(img[:,:,1]) #convierte la rebanada 2D en un arreglo 3D# Definir el colorcolor_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1)) #crea un arreglo de 1x1x3 - color magentacolor_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1)) #crea un arreglo de 1x1x3 - color verde# Cada píxel en la imagen se multiplica por el color.h1_magenta = h1_histone_3d * color_h1pila_green = pila_3d * color_pila# Fusionar los canales coloreadosmerged = np.clip(h1_magenta + pila_green,0,255)# Graficar los canales fusionados en el cuarto subplotaxs[3].imshow(merged)axs[3].axis('off')axs[3].set_title("Canales fusionados")# Ajustar espaciado entre los subplotsplt.tight_layout()Las dimensiones de las imágenes son 589 x 589 píxeles con 3 canales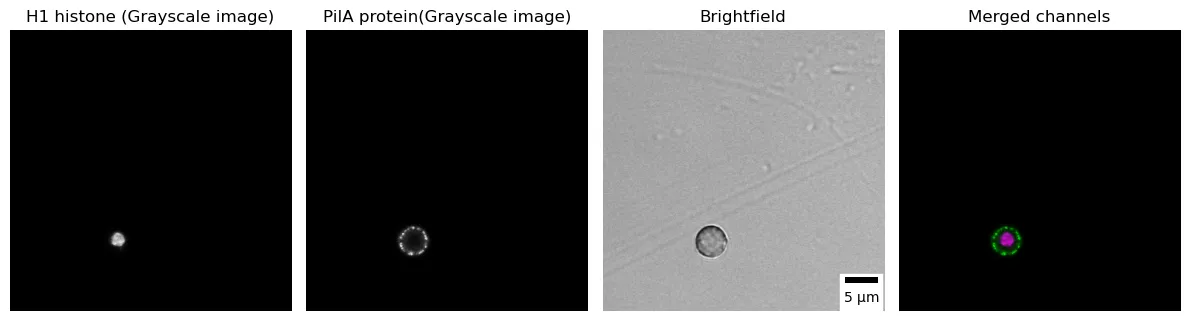
Imágenes 4D (x, y, canales y rebanadas Z)
Muy a menudo, en la microscopía confocal, capturamos “rebanadas” de imágenes en diferentes planos focales a lo largo del eje Z (una pila Z). Estas rebanadas se reconstruyen en una imagen 3D apilándolas juntas. Las pilas Z proporcionan información valiosa sobre la distribución espacial, morfología y relaciones de las estructuras dentro de la muestra en tres dimensiones. En el código a continuación, demostramos la visualización de la localización subcelular de PilA-GFP e Histone H1-mRFP en el conidiósporo de A. nidulans capturando cuatro “rebanadas” a lo largo del eje Z (En la práctica, la imagen a continuación representa un hiperpila, una composición 4D que consta de x, y, z y canales). Este enfoque nos permite estudiar la distribución y relaciones de estas proteínas en tres dimensiones.
# Leer la imagen usando scikit-image
img = sk.io.imread("3_channel_zstack_eisosome.tif")
# Imprimir las dimensiones de la imagen
print(f"Las dimensiones de las imágenes son {img.shape[1]} x {img.shape[2]} píxeles con {img.shape[3]} canales y {img.shape[0]} cortes")
# Separar los canales
h1_histone = img[:,:,:,0]
pila = img[:,:,:,1]
brightfield = img[:,:,:,2]
# Crear una figura con subplots
fig, axs = plt.subplots(3, 4, figsize=(12, 7))
# Graficar las imágenes individuales del canal H1 histone
for i in range(img.shape[0]):
axs[0][i].imshow(h1_histone[i,:,:],cmap="gray")
axs[0][i].axis('off')
axs[0][i].set_title(f"(Z = {i+1}) H1 histone")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[0, -1].add_artist(scalebar)
# Graficar las imágenes individuales del canal PilA protein
for i in range(img.shape[0]):
axs[1][i].imshow(pila[i,:,:],cmap="gray")
axs[1][i].axis('off')
axs[1][i].set_title(f"(Z = {i+1}) PilA protein")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[1, -1].add_artist(scalebar)
# Crear imágenes fusionadas de los canales H1 histone y PilA protein
for i in range(img.shape[0]):
h1_histone_3d = np.atleast_3d(h1_histone[i,:,:]) # convierte la imagen 2D a un arreglo 3D
pila_3d = np.atleast_3d(pila[i,:,:]) # convierte la imagen 2D a un arreglo 3D
# Definir el color
color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1)) # crea un arreglo 1x1x3 - color magenta
color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1)) # crea un arreglo 1x1x3 - color verde
h1_magenta = h1_histone_3d * color_h1
pila_green = pila_3d * color_pila
merged = np.clip(h1_magenta + pila_green,0,255)
axs[2][i].imshow(merged)
axs[2][i].axis('off')
axs[2][i].set_title(rf"(Z = {i+1}) H1 y PilA")
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[2, -1].add_artist(scalebar)
# Ajustar espaciado entre subplots
plt.tight_layout()
Las dimensiones de las imágenes son 589 x 589 píxeles con 3 canales y 4 cortes
Proyecciones
Una forma de visualizar un conjunto de imágenes Z es examinar cada corte individualmente, como se muestra en el código anterior. Sin embargo, este enfoque se vuelve complicado cuando se trata de múltiples cortes. Un método eficiente para resumir la información en un conjunto de imágenes Z es calcular una proyección Z [3]. Al realizar una proyección Z, podemos generar varios tipos de proyecciones en función de la pregunta específica que queremos responder. Por ejemplo, podemos obtener una proyección máxima, mínima, promedio o de desviación estándar. En una proyección máxima, se crea una imagen 2D que representa la intensidad máxima, capturando la característica más brillante, a lo largo de un eje específico, típicamente el eje Z. De manera similar, en las proyecciones de promedio, mínima y desviación estándar, generamos imágenes 2D que representan el promedio, mínimo o dispersión de las intensidades, respectivamente, a lo largo de un eje específico.
# Leer la imagen usando scikit-image
img = sk.io.imread("3_channel_zstack_eisosome.tif")
# Separar los canales
h1_histone = img[:,:,:,0]
pila = img[:,:,:,1]
brightfield = img[:,:,:,2]
# Crear una figura con subplots
fig, axs = plt.subplots(1, 4, figsize=(12, 7))
# Lista con los nombres de las proyecciones a mostrar
projections = ['Proyección mínima', 'Proyección media', 'Proyección máxima', 'Proyección de desviación estándar']
# Lista con los nombres de las proyecciones a calcular
np_projection = [np.min, np.mean, np.max, np.std]
# Calcular las proyecciones; mantener el mismo dtype en la salida
for i in range(len(projections)):
h1_histone_3d = np.atleast_3d(np_projection[i](h1_histone, axis=0).astype(h1_histone.dtype)) # convierte la imagen 2D a un arreglo 3D
pila_3d = np.atleast_3d(np_projection[i](pila, axis=0).astype(pila.dtype)) # convierte la imagen 2D a un arreglo 3D
color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1)) # crea un arreglo 1x1x3 - color magenta
color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1)) # crea un arreglo 1x1x3 - color verde
h1_magenta = h1_histone_3d * color_h1
pila_green = pila_3d * color_pila
merged = np.clip(h1_magenta + pila_green,0,255)
axs[i].imshow(merged)
scalebar = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right')
axs[i].add_artist(scalebar)
axs[i].axis('off')
axs[i].set_title(projections[i])

Imágenes 5D (x, y, canales, cortes Z y tiempo)
La imágenes confocales en 5D son una herramienta valiosa en biología, que ofrecen una visión sorprendente de los procesos dinámicos a nivel celular y subcelular. Estas imágenes comprenden cortes x, y, z y canales, junto con el tiempo, lo que permite la visualización y el análisis de los cambios temporales. En el siguiente código, demostramos la importancia de la imagen confocal en 5D presentando las proyecciones máximas de PilA-GFP y Histona H1-mRFP (imagen combinada) en cada punto de tiempo, siendo cada punto de tiempo aproximadamente 20 minutos.
# Leer la pila de imágenes 5D utilizando scikit-imageimg = sk.io.imread("3_channel_zstack_time_eisosome.tif")print(f"El conjunto de datos tiene {img.shape[2]} x {img.shape[3]} píxeles con {img.shape[4]} canales, {img.shape[1]} cortes y {img.shape[0]} puntos de tiempo")# Separar los canales h1_histone = img[:,:,:,:,0]pila = img[:,:,:,:,1]brightfield = img[:,:,:,:,2]# Crear una figura con subfigurasfig, axs = plt.subplots(6, 7, figsize=(19,14))# Calcular proyecciones (mantener el mismo tipo de datos en la salida) y crear imágenes combinadas para cada punto de tiempotiempo = 0for i in range (img.shape[0]): # Calcular la proyección máxima para el canal de histona H1 h1_histone_3d = np.atleast_3d(np.max(h1_histone[i,:,:,:], axis=0).astype(h1_histone.dtype)) # Calcular la proyección máxima para el canal de PilA pila_3d = np.atleast_3d(np.max(pila[i,:,:,:], axis=0).astype(pila.dtype)) # Calcular la proyección mínima para el canal de campo brillante brightfield_3d = np.atleast_3d(np.min(brightfield[i,:,:,:], axis=0).astype(brightfield.dtype)) # Definir los colores para cada canal color_h1 = np.asarray([1, 0, 1]).reshape((1, 1, -1)) #crea una matriz 1x1x3 - Color magenta color_pila = np.asarray([0, 1, 0]).reshape((1, 1, -1)) #crea una matriz 1x1x3 - Color verde color_brightfield = np.asarray([0.5, 0.5, 0.5]).reshape((1, 1, -1)) #crea una matriz 1x1x3 - Color gris # Crear la imagen combinada combinando los canales h1_magenta = h1_histone_3d * color_h1 pila_verde = pila_3d * color_pila brightfield_gris = brightfield_3d * color_brightfield # combinada = np.clip(h1_magenta + pila_verde + brightfield_gris,0,255) combinada = np.clip(h1_magenta + pila_verde,0,255)# Mostrar la imagen combinada en la subfigura correspondiente axs[i // 7, i % 7].imshow(combinada) axs[i // 7, i % 7].axis('off') axs[i // 7, i % 7].set_title(f"{tiempo} min") escala = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right') axs[5, 1].add_artist(escala) tiempo += 20plt.tight_layout()plt.show()El conjunto de datos tiene 589 x 589 píxeles con 3 canales, 4 cortes y 37 puntos de tiempo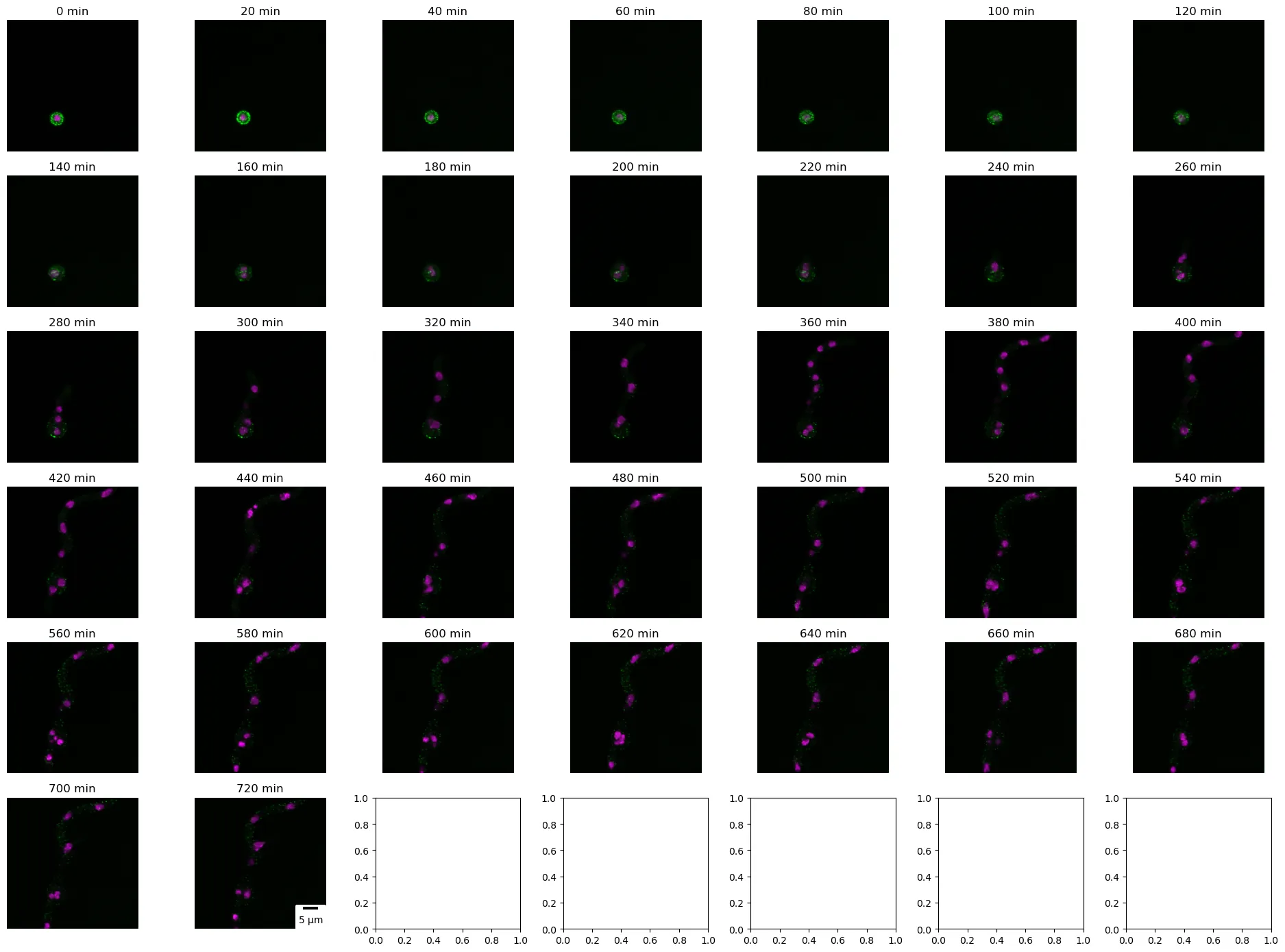
A continuación se muestra una imagen de campo brillante del hongo A. nidulans en diferentes puntos de tiempo.
# Leer la pila de imágenes 5D utilizando scikit-imageimg = sk.io.imread("3_channel_zstack_time_eisosome.tif")# Separar los canales h1_histone = img[:,:,:,:,0]pila = img[:,:,:,:,1]brightfield = img[:,:,:,:,2]# Crear una figura con subfigurasfig, axs = plt.subplots(6, 7, figsize=(19,14))# Calcular proyecciones (mantener el mismo tipo de datos en la salida) y crear imágenes combinadas para cada punto de tiempotiempo = 0for i in range (img.shape[0]):# Seleccionar un corte del canal de campo brillante brightfield_2_slice = brightfield[i,2,:,:]# Mostrar el segundo corte en la subfigura correspondiente axs[i // 7, i % 7].imshow(brightfield_2_slice, cmap="gray") axs[i // 7, i % 7].axis('off') axs[i // 7, i % 7].set_title(f"{tiempo} min") escala = ScaleBar(0.0721501, 'um', length_fraction=0.2, height_fraction=0.02, location='lower right') axs[5, 1].add_artist(escala) tiempo += 20plt.tight_layout()plt.show()

Conclusiones
En este tutorial, nos adentraremos en el proceso de lectura y visualización de imágenes intrincadas de microscopio de luz con diversas dimensiones, incluyendo 2D, 3D, 4D y 5D. Aunque matplotlib es una biblioteca poderosa para la visualización de datos, puede que no sea la elección más adecuada para visualizar imágenes 3D, 4D y 5D, como las imágenes microscópicas. Si bien matplotlib puede manejar imágenes 2D de manera efectiva, la visualización de imágenes de mayor dimensionalidad puede ser desafiante y limitada. Sin embargo, existen soluciones:
- Microfilm [9], [10], que simplifica la representación de imágenes de múltiples canales
- y Napari [8], una popular biblioteca de Python diseñada específicamente para la visualización y exploración de datos científicos multidimensionales, incluyendo imágenes microscópicas. Napari proporciona una interfaz interactiva y versátil que va más allá de las capacidades de matplotlib.
En nuestro próximo tutorial, profundizaremos en Napari y exploraremos sus características en detalle para comprender mejor sus capacidades para visualizar imágenes de mayor dimensionalidad.
He preparado un Jupyter Notebook para acompañar esta publicación de blog, el cual se puede ver en mi GitHub.

Referencias:
[1] C. T. Rueden y K. W. Eliceiri, “Visualization approaches for multidimensional biological image data,” BioTechniques, vol. 43, no. 1S, pp. S31-S36, Jul. 2007, doi: 10.2144/000112511.
[2] J. B. Pawley, “Points, Pixels, and Gray Levels: Digitizing Image Data,” en Handbook Of Biological Confocal Microscopy, J. B. Pawley, Ed., Boston, MA: Springer US, 2006, pp. 59-79. doi: 10.1007/978-0-387-45524-2_4.
[3] P. Bankhead, “Introduction to Bioimage Analysis – Introduction to Bioimage Analysis.” https://bioimagebook.github.io/index.html (accedido el 29 de junio de 2023).
[4] J. Johnson, “Not seeing is not believing: improving the visibility of your fluorescence images,” Mol. Biol. Cell, vol. 23, no. 5, pp. 754-757, Mar. 2012, doi: 10.1091/mbc.e11-09-0824.
[5] I. Vangelatos, K. Roumelioti, C. Gournas, T. Suarez, C. Scazzocchio y V. Sophianopoulou, “Eisosome Organization in the Filamentous Ascomycete Aspergillus nidulans,” Eukaryot. Cell, vol. 9, no. 10, pp. 1441-1454, Oct. 2010, doi: 10.1128/EC.00087-10.
[6] A. Athanasopoulos, C. Gournas, S. Amillis y V. Sophianopoulou, “Characterization of AnNce102 and its role in eisosome stability and sphingolipid biosynthesis,” Sci. Rep., vol. 5, no. 1, Dec. 2015, doi: 10.1038/srep15200.
[7] A. P. Mela y M. Momany, “Difusión internuclear de la histona H1 dentro de los compartimentos celulares de Aspergillus nidulans”, PloS One, vol. 13, no. 8, p. e0201828, 2018, doi: 10.1371/journal.pone.0201828.
[8] “napari: un visor rápido e interactivo para imágenes multidimensionales en Python – napari.” https://napari.org/stable/ (consultado el 04 de julio de 2023).
[9] G. Witz, “microfilm”. 27 de junio de 2023. Consultado: 14 de julio de 2023. [En línea]. Disponible en: https://github.com/guiwitz/microfilm
[10] “Cuadernos de análisis de imágenes biológicas – Cuadernos de análisis de imágenes biológicas.” https://haesleinhuepf.github.io/BioImageAnalysisNotebooks/intro.html (consultado el 14 de julio de 2023).
We will continue to update Zepes; if you have any questions or suggestions, please contact us!
Was this article helpful?
93 out of 132 found this helpful
Related articles
- Guía introductoria completa sobre el reconocimiento de voz a texto con Transformers
- ¿Cómo cambiar tu carrera de Marketing a Ciencia de Datos?
- Reduciendo la huella de carbono en el entrenamiento de IA mediante la optimización
- Investigadores de IA de Google presentan Pic2Word Un nuevo enfoque para la recuperación de imágenes compuestas sin disparo (ZS-CIR)
- Conoce a PoisonGPT Un método de IA para introducir un modelo malicioso en una cadena de suministro de LLM de otra manera confiable
- Informe de la OCDE el 27% de los empleos están en alto riesgo debido a la IA
- La IA está ayudando a los pacientes de ELA a preservar su voz






